はじめに
Databricks環境下でオープンソースプラットフォームである MLflow を利用します。
実験データの統合・トラッキング方法を解説します。
また、本記事は Python3 を想定しています。
この記事の概要
- Databricks 環境で MLfow を使うことできること
- MLflow の使い方
- サンプルデータを用いた実験の情報をノートブックに統合・トラッキング方法
Databricksのマネージド型MLflow
オープンソースであるMLflowはあらゆる ML ライブラリや言語、デプロイメントツールで動作するように設計されていますが、
自前で実験のトラッキング用のサーバーを用意する必要があります。
Databricks環境下では
- MLflowはマネージド型のサービスとして利用可能。
- トラッキング用のサーバーを別途用意する必要なし。
- 実験のトラッキング情報をノートブックに統合して管理可能。
MLflowの使い方
Databricks上で使っているクラスターがRuntime MLの場合は最初から入っています。
それ以外の場合はMLflowをインストールする必要です。
|
1 2 3 |
dbutils.library.installPyPI("mlflow") dbutils.library.restartPython() |
上記コマンドでインストールできます。そしてインポートします。
|
1 2 |
import mlflow |
MLflowでは
- トラッキング開始のモジュールを呼び出してトラッキングを開始
- 実験のパラメータやログを記録するモジュールで記録
- トラッキング終了のモジュールで一つの実験トラッキングが終了 という形になります。
終了し忘れ防止のためにもwithを使うのがいいと思います。
実装部分のコードのイメージとしては以下の通りです。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
with mlflow.start_run(): # 実験トラッキング開始 # 実験の処理 # ログとパラメータなどの記録 mlflow.log_param("a", a) mlflow.log_metric("rmse", rmse) # モデルの記録 mlflow.sklearn.save_model(b, modelpath) # 実験中に出力した画像などの保存 mlflow.log_artifact("sample.png") |
パラメータ以外にもモデルの記録や実験中に出力した画像などもトラッキング先に保存できます。
モデルの記録には、該当するライブラリのインストールが別途必要です。
例)scikit-learnのモデル → mlflow.sklearn
実装
実際に実装してノートブック上でトラッキングしてみます。
サンプルデータセットと使用モデル
scikit-learnの糖尿病データセットを使います。
カラムの説明などはこちらにあります。
https://scikit-learn.org/stable/datasets/index.html#diabetes-dataset
今回のモデル作成にはElasticNet線形回帰モデルを使います。
調整パラメータとしてalpha、l1_ratioがあります。
ElasticNetについてはこちらの説明がわかりやすかったです。
https://aizine.ai/ridge-lasso-elasticnet/
セットアップ
各種ライブラリのインポートとサンプルデータセットの読み込み、データフレームの作成を行います。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 |
# 必要なライブラリの読み込み import os import warnings import sys import pandas as pd import numpy as np from itertools import cycle import matplotlib.pyplot as plt from sklearn.metrics import mean_squared_error, mean_absolute_error, r2_score from sklearn.model_selection import train_test_split from sklearn.linear_model import ElasticNet from sklearn.linear_model import lasso_path, enet_path from sklearn import datasets # mlflowのインポート import mlflow import mlflow.sklearn # 糖尿病データセットのロード diabetes = datasets.load_diabetes() X = diabetes.data y = diabetes.target # データフレームの作成 Y = np.array([y]).transpose() d = np.concatenate((X, Y), axis=1) cols = ['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6', 'progression'] data = pd.DataFrame(d, columns=cols) |
結果処理部分の実装
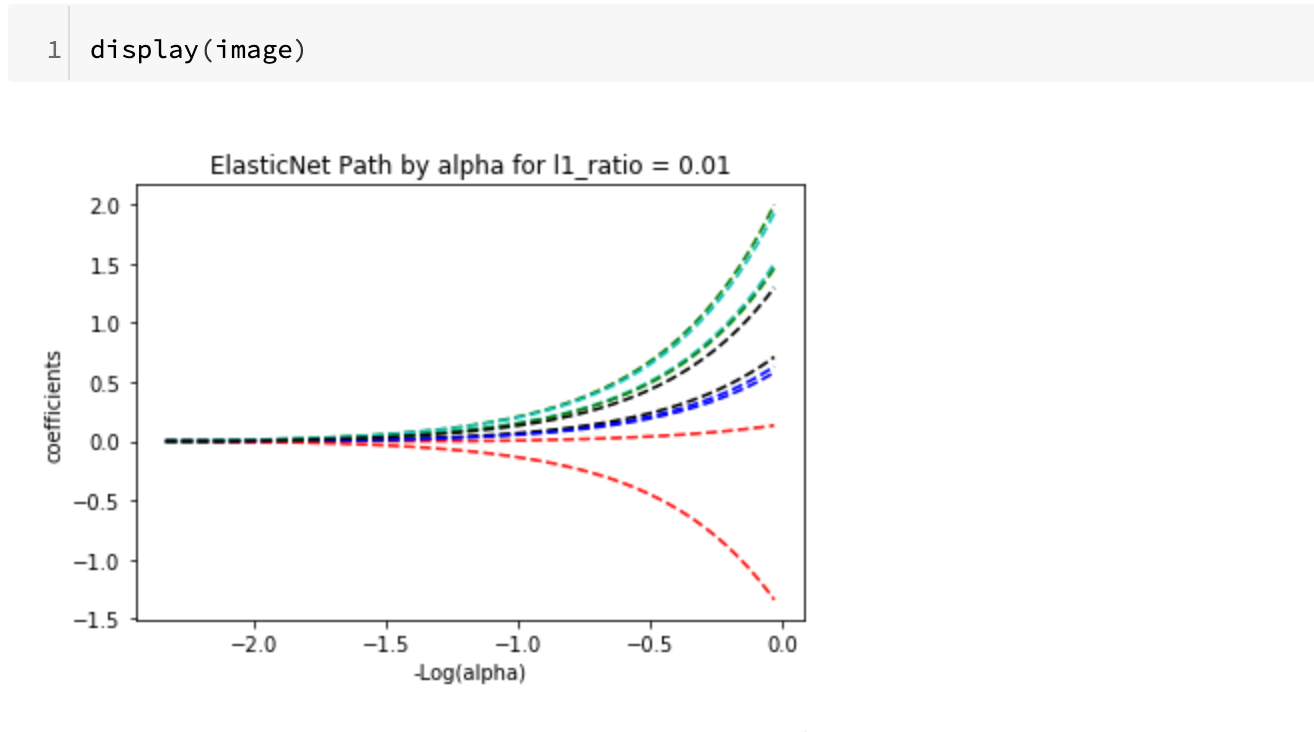
ElasticNetで回帰モデルを作ったときの説明変数の各係数をプロットして、それを画像としてドライバノードに保存する処理を定義しています。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 |
def plot_enet_descent_path(X, y, l1_ratio): # パスの長さ(alpha_min / alpha_max)の設定 eps = 5e-3 # imageのグローバル宣言 global image print("Computing regularization path using ElasticNet.") alphas_enet, coefs_enet, _ = enet_path(X, y, eps=eps, l1_ratio=l1_ratio, fit_intercept=False) # 結果の表示 fig = plt.figure(1) ax = plt.gca() colors = cycle(['b', 'r', 'g', 'c', 'k']) neg_log_alphas_enet = -np.log10(alphas_enet) for coef_e, c in zip(coefs_enet, colors): l1 = plt.plot(neg_log_alphas_enet, coef_e, linestyle='--', c=c) plt.xlabel('-Log(alpha)') plt.ylabel('coefficients') title = 'ElasticNet Path by alpha for l1_ratio = ' + str(l1_ratio) plt.title(title) plt.axis('tight') image = fig # 画像の保存 fig.savefig("ElasticNet-paths.png") # plotのクローズ plt.close(fig) # Return images return image |
実験処理部分の実装
alphaとl1_ratioを指定してモデルのトレーニングを行います。上で定義したplot_enet_descent_pathを呼び出してトラッキング先にログや画像を保存します。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 |
def train_diabetes(data, in_alpha, in_l1_ratio): # メトリクスの評価 def eval_metrics(actual, pred): rmse = np.sqrt(mean_squared_error(actual, pred)) mae = mean_absolute_error(actual, pred) r2 = r2_score(actual, pred) return rmse, mae, r2 warnings.filterwarnings("ignore") np.random.seed(40) # データセットの分割 train, test = train_test_split(data) # 正解ラベルの分割 train_x = train.drop(["progression"], axis=1) test_x = test.drop(["progression"], axis=1) train_y = train[["progression"]] test_y = test[["progression"]] if float(in_alpha) is None: alpha = 0.05 else: alpha = float(in_alpha) if float(in_l1_ratio) is None: l1_ratio = 0.05 else: l1_ratio = float(in_l1_ratio) # mlflowの実装部分 # mlflow.start_run()の引数の値を空にすることでノートブックに統合されます with mlflow.start_run(): lr = ElasticNet(alpha=alpha, l1_ratio=l1_ratio, random_state=42) lr.fit(train_x, train_y) predicted_qualities = lr.predict(test_x) (rmse, mae, r2) = eval_metrics(test_y, predicted_qualities) # ElasticNetモデルのメトリクスの表示 print("Elasticnet model (alpha=%f, l1_ratio=%f):" % (alpha, l1_ratio)) print(" RMSE: %s" % rmse) print(" MAE: %s" % mae) print(" R2: %s" % r2) # ログの保存 mlflow.log_param("alpha", alpha) mlflow.log_param("l1_ratio", l1_ratio) mlflow.log_metric("rmse", rmse) mlflow.log_metric("r2", r2) mlflow.log_metric("mae", mae) mlflow.sklearn.log_model(lr, "model") modelpath = "/dbfs/mlflow/test_diabetes/model-%f-%f" % (alpha, l1_ratio) mlflow.sklearn.save_model(lr, modelpath) # plot_enet_descent_pathの呼び出し image = plot_enet_descent_path(X, y, l1_ratio) # 出力画像の保存 mlflow.log_artifact("ElasticNet-paths.png") |
実験
調整パラメータを与えて実験を行います。
|
1 2 3 |
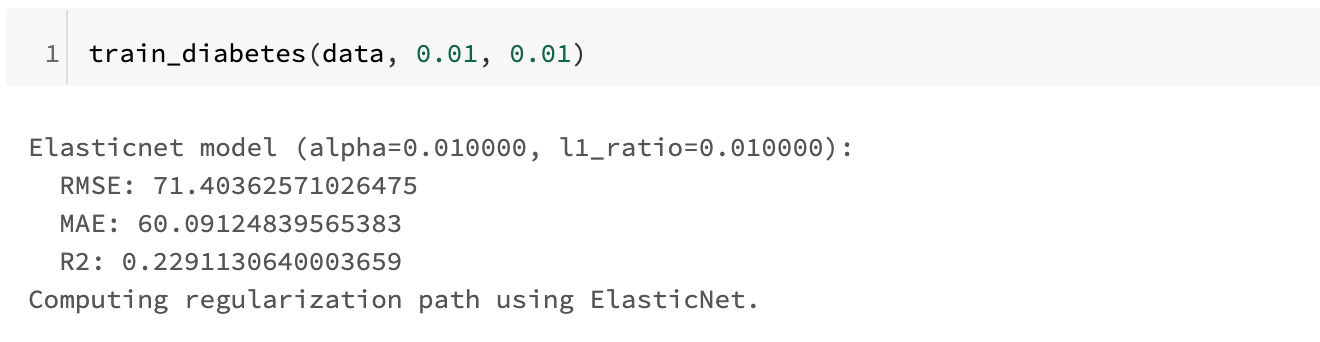
# alpha = 0.01, l1_ratio = 0.01として実験 train_diabetes(data, 0.01, 0.01) |
出力結果は次のようになりました。
いくつかパラメータを変えて実験してみます。(合計4パターンほどやってみました)
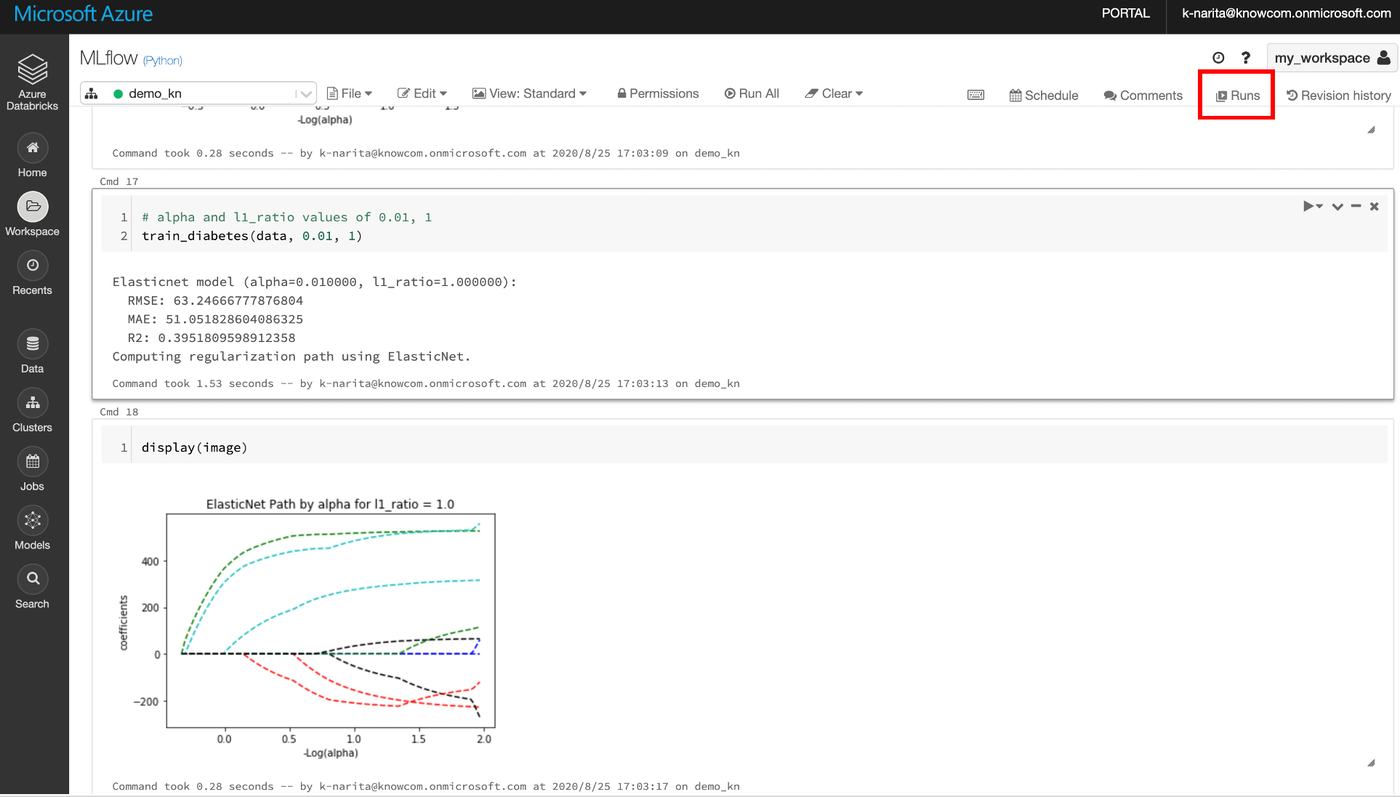
実験が終わったら右上のあたりに[Runs]と書かれた部分を押します。
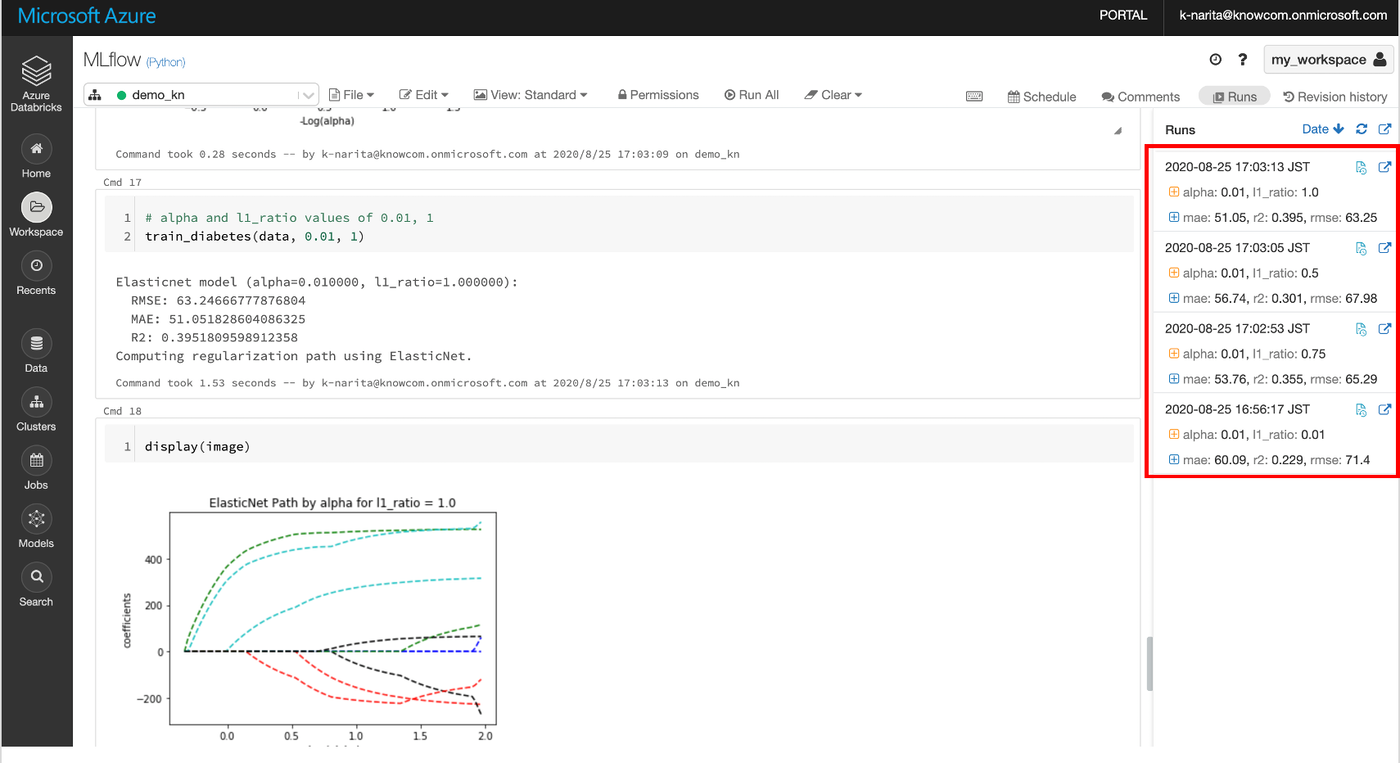
設定したパラメータの値と出力されたメトリクスが実験ごとに記録されているのが確認できます。
おわりに
今回はノートブック上でモデルのトレーニング結果をノートブックに統合・トラッキングする方法について紹介しました。
Databricks では UI 上でこちらの実験データを比較ができるようになっています。
続編として、UI 上のモデル管理について書きたいと思います。
次回もお楽しみに!